您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
如何進行WES的CNV分析,相信很多沒有經驗的人對此束手無策,為此本文總結了問題出現的原因和解決方法,通過這篇文章希望你能解決這個問題。
基于全基因組數據來檢測CNV是非常有效的一個手段,然而全基因組的成本還是挺高的。全外顯子組在檢測SNP方面已經比較成熟,考慮到外顯子上的變異可能更具有致病性,科研人員也希望通過檢測外顯子上的CNV來實現一個高效,經濟的CNV檢測,很多的軟件被開發用于WES的CNV分析。
CNV區域的長度可能橫跨了多個外顯子或者基因,斷裂點位于外顯子以外的位置,所以全基因組分析中Read-pair, split-read的策略無法應用到WES的CNV分析中,只能通過read-depth的策略來進行分析。
然而和全基因組不同,全外顯子靶向捕獲了基因組的外顯子區域,考慮到GC含量,序列捕獲等系統誤差,其測序深度的分布和CNV之間的相關性更加復雜,建模衡量的難度更大,所以之前適用于WGS分析的CNV檢測軟件很多都不可以用于WES的分析。
為了有效減少系統誤差的影響,提高CNV檢測的準確率,很多WES的分析軟件都會需要一個對照樣本,將對照樣本和測試樣本進行比較來識別二者間差異的地方,從而回避系統誤差帶來的影響。同樣的protocol意味著同樣的系統誤差,而二者直接還存在的差異就是由于樣本本身的差異引起的了,這就是對照樣本的作用。所以WES的CNV檢測經典的用處就是檢測體細胞CNV,即SCNA變異,提供配對的癌和癌旁樣本來進行分析。
在以下文獻中,詳細列舉了幾種外顯子CNV檢測的軟件
https://academic.oup.com/bib/article/16/3/380/245577
根據是否需要對照樣本分成以下3大類
paired data, 需要配對的對照樣本
pooled data, 不需要對照樣本
paired and pooled data, 兩種策略都可以
軟件列表如下
ExomeCNV
Varscan2
Control-Freec
exome2cnv
PropSeg
2. pooled data
軟件列表如下
condex
exomeCOPY
cn.mops
conifer
ExomeDepth
XHMM
ExoCNVTest
Excavator
軟件列表如下
contar
ADTEx
FishingCNV
該文章發表于2014年,在之后又陸續發表了很多新工具,比如excavator, 2016年發表在Nucleic Acids Research上的文章介紹了excavator2進行CNV分析的強大之處,鏈接如下
https://academic.oup.com/nar/article/44/20/e154/2607979
不同工具算法模型都各不相同,各有優劣,在2014年發表的一篇文章對多個軟件進行了評估,標題如下

在文章中,列舉了很多CNV分析的軟件,示意如下
最終選取了以下4款軟件來進行評估
XHMM
CoNIFER
ExomeDepth
CONTRA
從以下多個方面進行了評估
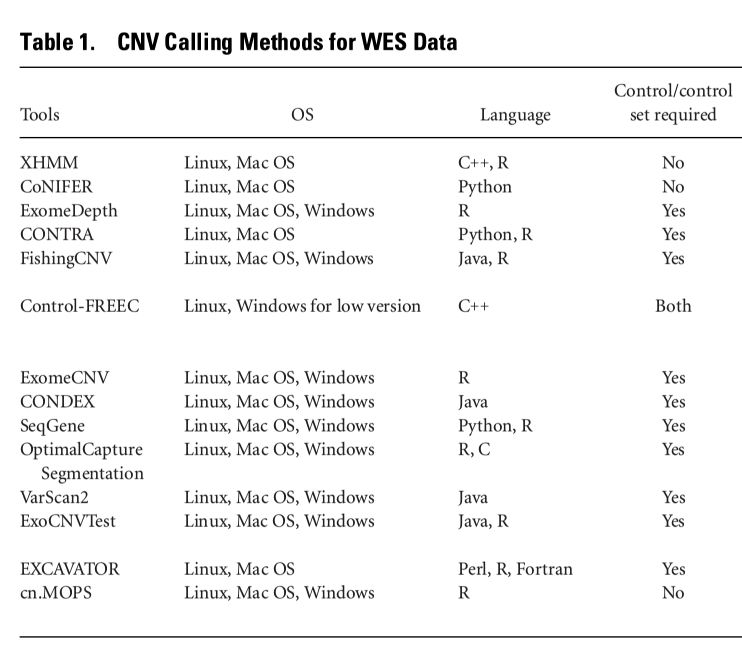
不同軟件檢測到的CNV長度分布不同,結果統計如下
CNV的長度可以從幾十bp跨越到幾Mb的范圍,通常認為小于300bp和長度在6kb左右的CNV應該是數量最多的。WES的CNV檢測工具都是基于read-depth算法,采用滑動窗口的方法,窗口越大,最終鑒定出來的CNV可信度越高,所以在檢測小片段的CNV方面,能力較差。
從統計結果可以看出,Conifer沒有鑒定出1kb以下的CNV, 因為這款軟件要求CNV至少需要覆蓋3個exon區域,XHMM和ExomeDepth則可以同時檢測小片段和大片段的CNV, CONTRA檢測出來的數量過多,是由于其校正read-depthh的算法過于敏感,所以鑒定出來的CNV過多,在檢測小于1kb的小片段CNV時,比較適合。
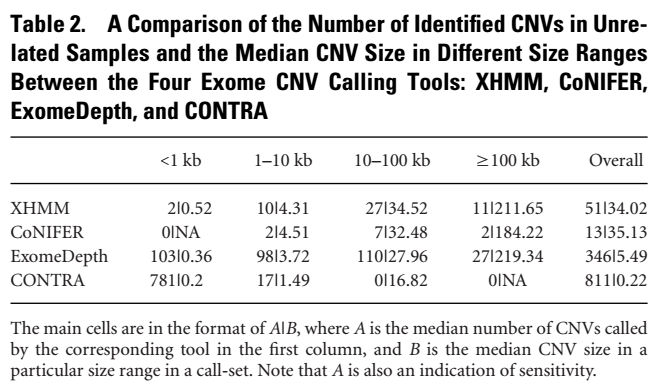
不同軟件鑒定到的CNV的數量和類型展示如下
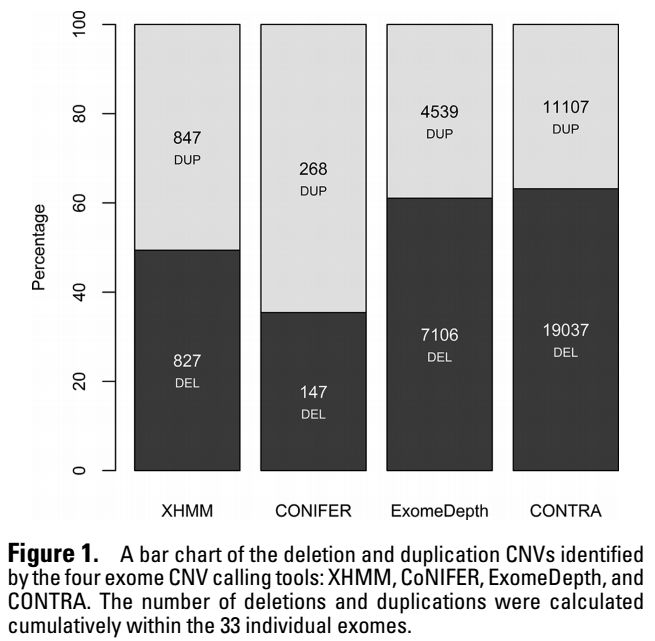
采用了cnvnator和ERDS兩款軟件對WGS數據進行CNV檢測,然后和WES的結果進行一致性分析,以exon為單位進行評估,當一個exon 50%以上的區域落在CNV區域時進行計算,比較不同軟件檢測到的exon和WGS數據exon的overlap情況,結果如下

盡管都很低,但是很明顯ExomeDepth overlap率最高,接下來是XHMM。
利用1000G項目中在人群中頻率大于5%的cnvs作為common cnv, 采用上述的方法評估不同軟件和common cnv的一致性,結果和WGS一致,也是ExomeDepth最高,XHMM次之。
通常情況下denovo CNV的概率是非常低的,將denovo CNV作為Mendelian Error Rate的指標,對個體及其雙親同時進行CNV分析,評估denovo cnv的頻率,結果如下

每個軟件不符合孟德爾遺傳的CNV比例都很高,conifer最高,而CONTRA最低。
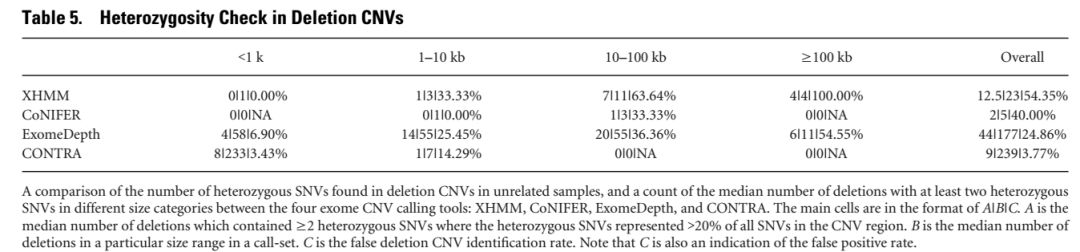
對于deletion CNV而言,其染色體區域只剩下一份拷貝,在該區域內的SNV必然為純合性的,所以將包含了雜合SNV的CNV區域作為假陽性的結果,考慮到SNP分型的準確率,將同時滿足以下兩個條件的缺失區域定義為假陽性的結果
包含了2個以上的雜合SNP
20%以上的SNP位點為雜合
拷貝數缺失的假陽性統計結果如下
基于exon水平來統計不同軟件之間的一致性,結果如下所示
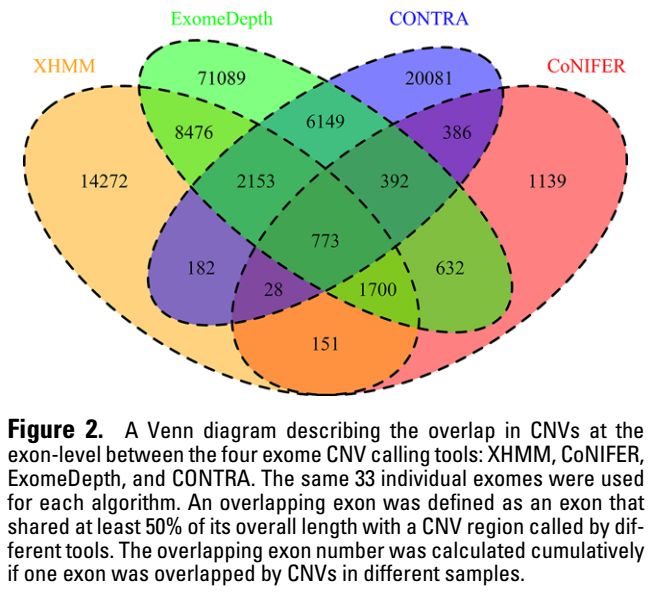
綜合以上6個指標來看,沒有哪個軟件是全面優于其他軟件的,在不同指標上,不同軟件各有優劣。
在進行WES的CNV檢測時,基于一款軟件的結果很難兼顧靈敏度和特異性,最好的方法還是結合多款軟件的結果進行判斷。
看完上述內容,你們掌握如何進行WES的CNV分析的方法了嗎?如果還想學到更多技能或想了解更多相關內容,歡迎關注億速云行業資訊頻道,感謝各位的閱讀!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。