您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
本篇文章給大家分享的是有關PennCNV如何利用SNP芯片檢測CNV,小編覺得挺實用的,因此分享給大家學習,希望大家閱讀完這篇文章后可以有所收獲,話不多說,跟著小編一起來看看吧。
通過SNP芯片來檢測CNV,對應的分析軟件有很多,PennCNV就是其中之一,該軟件通過隱馬可夫模型來檢測CNV, 官網如下
http://penncnv.openbioinformatics.org/en/latest/
數據分析的pipeline示意如下
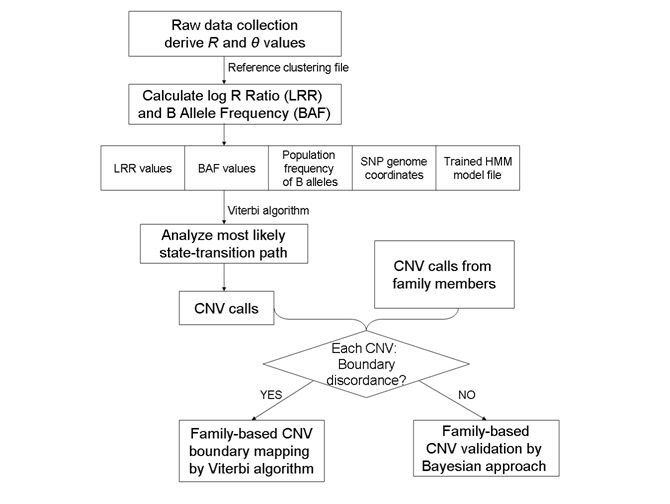
首先我們需要準備signal intensity file, 該文件用來記錄每個位點對應的LRR和BAF兩個統計值,有兩種格式,第一種格式示意如下
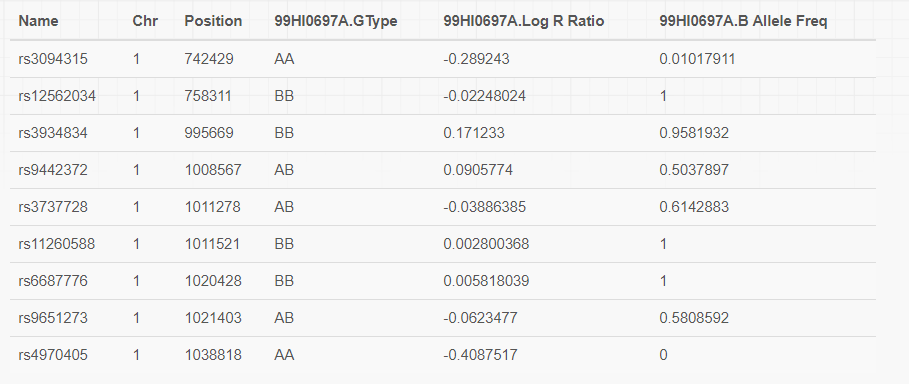
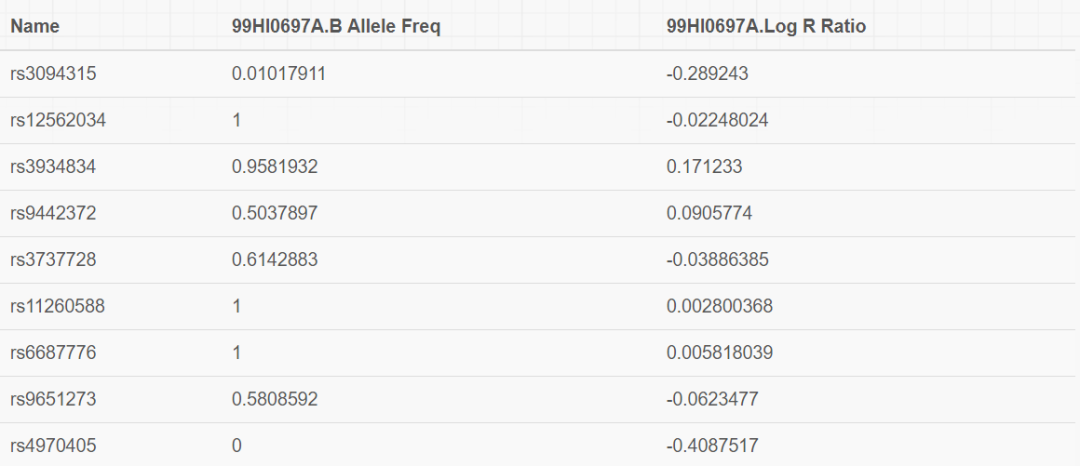
\t分隔的6列,第一列為SNP的名稱,第二列為snp位點所在的染色體名稱,第三列為snp位點所在的染色體位置,第四列為該位點的分型結果,第五列為LRR統計值,第六列為BAF統計值,對于cnv calling而言,最重要的是Name, LRR, BAF這3列的值,所以該文件還可以有第二種格式,示意如下
其次需要芯片平臺對應的HMM和PFB文件,在軟件的安裝目錄自帶了部分芯片對應的這兩種文件。PFB是population frequency of B allel的縮寫,本質是每個SNP位點的MAF, 同時還提供了染色體位置的注釋信息,內容示意如下
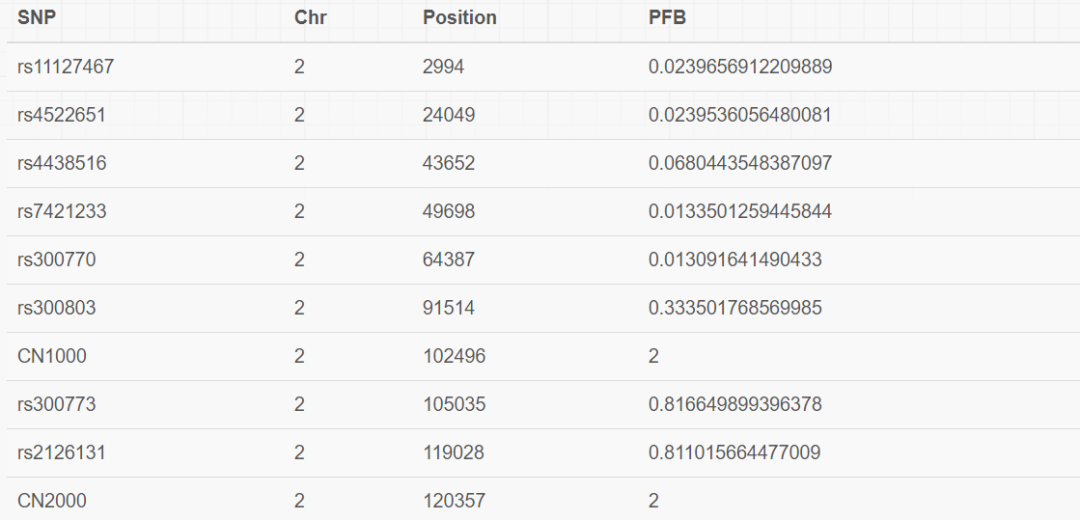
對于SNP芯片上集成的非SNP探針,約定其PFB的值為2。需要注意的是,只有該文件中記錄的位點才會用于CNV calling的分析,當我們需要篩選位點時,只需要在該文件中進行過濾即可。HMM文件是軟件自帶的,提供了不同拷貝數之間的轉移概率。準備好輸入文件之后,就可以進行分析了,常見的分析步驟如下
對于獨立樣本,檢測CNV的命令如下
detect_cnv.pl \
-test \
-hmm lib/hh650.hmm \
-pfb lib/hh650.hg18.pfb \
sample.txt \
-log sample.log \
-out sample.rawcnv輸出內容如下所示
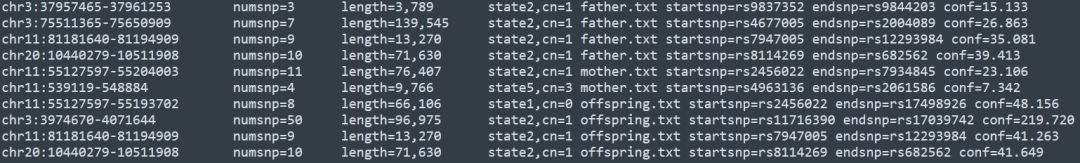
第一列為CNV的染色體區域,第二列為該CNV區域包含的SNP位點數目,第三列為CNV區域的長度,第四列中cn表示該CNV區域的拷貝數,后面依次是樣本對應的輸入文件,起始和終止的snp name, 打分值。
常見的篩選策略如下
根據長度和包含的位點數等統計指標進行過濾
根據染色體區域進行篩選,過濾掉位于特殊區域的CNV,比如著絲粒,端粒等區域
根據長度過濾的命令如下
filter_cnv.pl \
-numsnp 10 \
-length 50k \
sampleall.rawcnv過濾掉包含的snp位點數少于10個,長度小于50kb的CNV區域。
通常會對CNV區域進行以下注釋
overlap的基因
上下游最鄰近的基因
overlap的功能元件
命令如下
scan_region.pl \
sampleall.rawcnv \
hg19_refGene.txt \
-refgene \
-reflink hg19_refLink.txt \
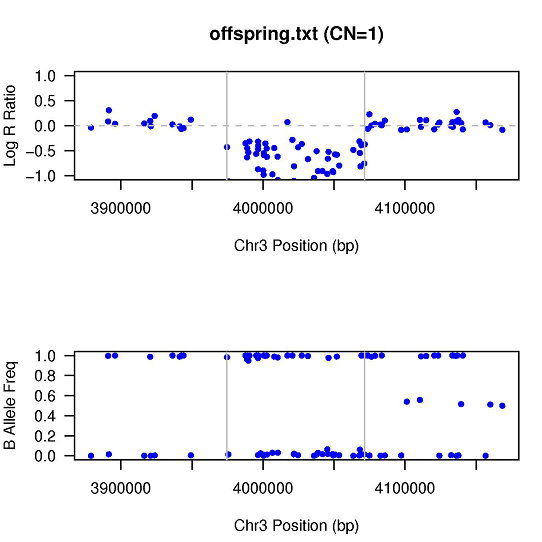
> sampleall.cnv.hg19對CNV區域內SNP位點的LRR和BAF值進行可視化,命令如下
visualize_cnv.pl \
-format plot \
-signal sample.txt \
sampleall.rawcnv結果示意如下
該軟件還有很多高級的用法,比如家系樣本的CNV檢測,case/control的關聯分析等等。
以上就是PennCNV如何利用SNP芯片檢測CNV,小編相信有部分知識點可能是我們日常工作會見到或用到的。希望你能通過這篇文章學到更多知識。更多詳情敬請關注億速云行業資訊頻道。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。