您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
本篇內容主要講解“R語言怎么使用cgdsr包獲取TCGA數據”,感興趣的朋友不妨來看看。本文介紹的方法操作簡單快捷,實用性強。下面就讓小編來帶大家學習“R語言怎么使用cgdsr包獲取TCGA數據”吧!
眾所周知,TCGA數據庫是目前最綜合全面的癌癥病人相關組學數據庫,包括的測序數據有:
DNA Sequencing
miRNA Sequencing
Protein Expression
mRNA Sequencing
Total RNA Sequencing
Array-based Expression
DNA Methylation
Copy Number
知名的腫瘤研究機構都有著自己的TCGA數據庫探索工具,比如:
Broad Institute FireBrowse portal, The Broad Institute
cBioPortal for Cancer Genomics, Memorial Sloan-Kettering Cancer Center
TCGA Batch Effects, MD Anderson Cancer Center
Regulome Explorer, Institute for Systems Biology
Next-Generation Clustered Heat Maps, MD Anderson Cancer Center
其中cBioPortal更是被包裝到R包里面
這里介紹如何使用R語言的cgdsr包來獲取任意TCGA數據。
cgdsr包:R語言工具包,可以下載TCGA數據。
DT包:data.table包,簡稱DT包,是R語言中的數據可視化工具包。DT包可以將Javascript中的方法運用到R中,也能將矩陣或者數據表在網頁中可視化為表格,以及其它的一些功能。
> setwd("C:/Users/YLAB/Documents/R/win-library/4.1/")
> install.packages("R.methodsS3_1.8.1.zip",repos=NULL)#安裝
> install.packages("R.oo_1.24.0.zip",repos=NULL)#安裝
> install.packages("data.table")
> BiocManager::install("cgdsr", force = TRUE)#安裝
> library(cgdsr)
> library(DT)
#創建一個cgdsr對象
> mycgds <- CGDS("http://www.cbioportal.org/")
#檢查下載是否成功,如果是FAILED就是沒成功。
> test(mycgds)
getCancerStudies... OK
getCaseLists (1/2) ... OK
getCaseLists (2/2) ... OK
getGeneticProfiles (1/2) ... OK
getGeneticProfiles (2/2) ... OK
getClinicalData (1/1) ... OK
getProfileData (1/6) ... OK
getProfileData (2/6) ... OK
getProfileData (3/6) ... OK
getProfileData (4/6) ... OK
getProfileData (5/6) ... OK
getProfileData (6/6) ... OK
all_TCGA_studies <- getCancerStudies(mycgds)
> DT::datatable(all_TCGA_studies)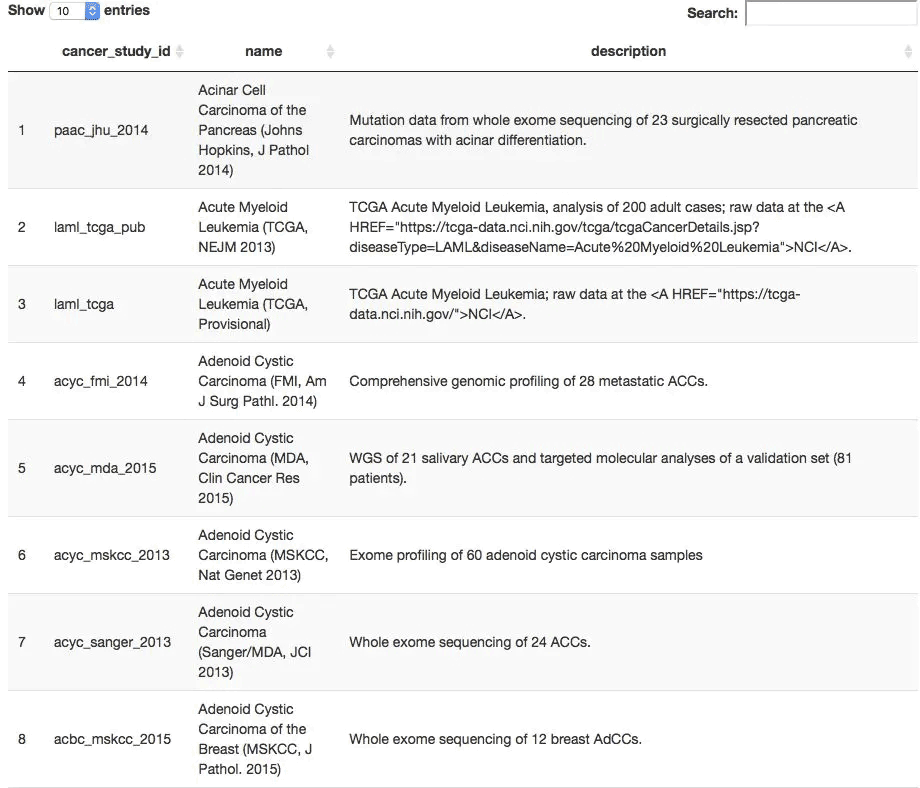
上表的cancer_study_id其實就是數據集的名字,我們任意選擇一個數據集,比如stad_tcga_pub ,可以查看它里面有多少種樣本列表方式。
stad2014 <- "stad_tcga_pub" ## 獲取在stad2014數據集中有哪些表格(每個表格都是一個樣本列表) all_tables <- getCaseLists(mycgds, stad2014) dim(all_tables) ## 共6種樣本列表方式 [1] 6 5 DT::datatable(all_tables[,1:3])
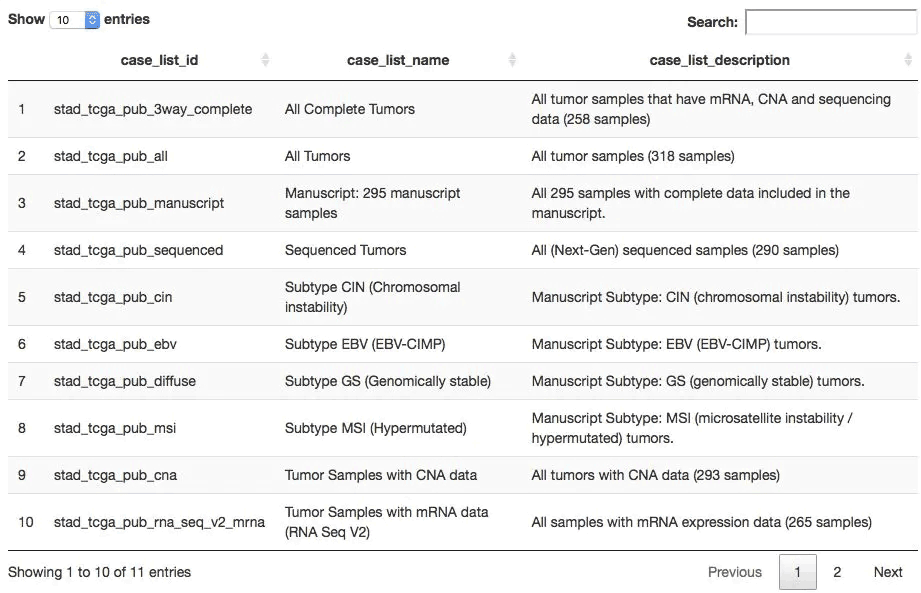
查看任意數據集的數據形式
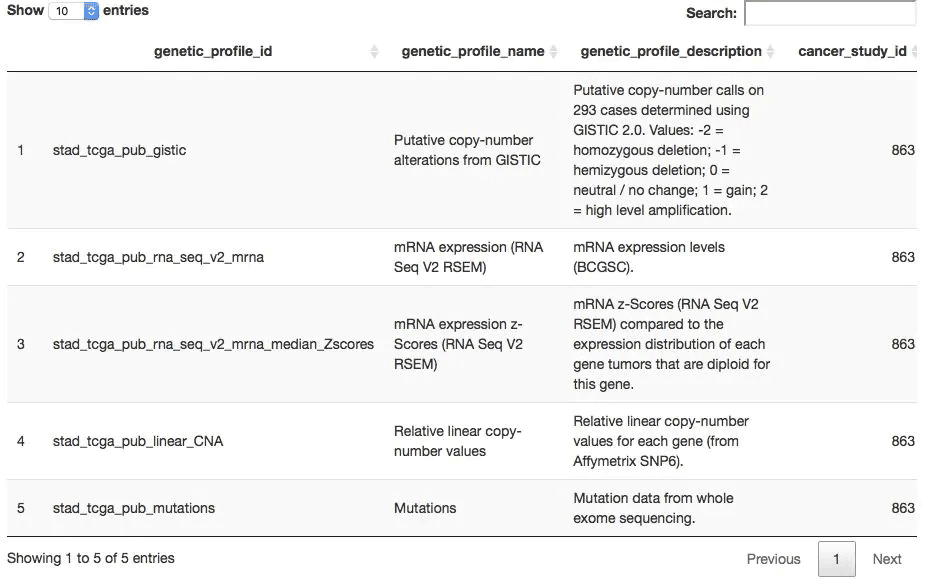
## 而后獲取可以下載哪幾種數據,一般是mutation,CNV和表達量數據 all_dataset <- getGeneticProfiles(mycgds, stad2014) DT::datatable(all_dataset, extensions = 'FixedColumns', options = list( #dom = 't', scrollX = TRUE, fixedColumns = TRUE ))
一般來說,TCGA的一個項目數據就幾種,如下:
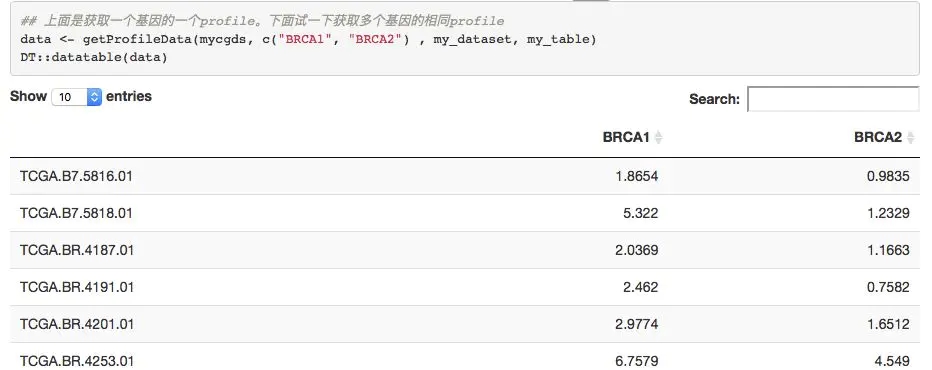
my_dataset <- 'stad_tcga_pub_rna_seq_v2_mrna' my_table <- "stad_tcga_pub_rna_seq_v2_mrna" BRCA1 <- getProfileData(mycgds, "BRCA1", my_dataset, my_table) dim(BRCA1) [1] 265 1
樣本個數差異很大,不同癌癥熱度不一樣。
## 如果我們需要繪制survival curve,那么需要獲取clinical數據 clinicaldata <- getClinicalData(mycgds, my_table) DT::datatable(clinicaldata, extensions = 'FixedColumns', options = list( #dom = 't', scrollX = TRUE, fixedColumns = TRUE ))
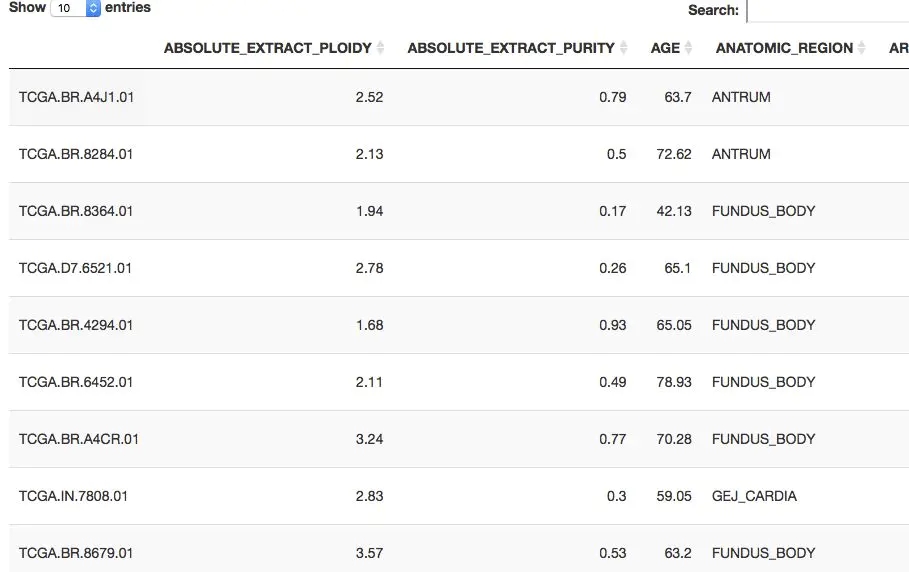
只需要根據癌癥列表選擇自己感興趣的研究數據集即可,然后選擇好感興趣的數據形式及對應的樣本量。就可以獲取對應的信息:
library(cgdsr)
library(DT)
mycgds <- CGDS("http://www.cbioportal.org")
##mycancerstudy = getCancerStudies(mycgds)[25,1]
mycancerstudy = 'brca_tcga' getCaseLists(mycgds,mycancerstudy)[,1]
## [1] "brca_tcga_3way_complete" "brca_tcga_all"
## [3] "brca_tcga_protein_quantification" "brca_tcga_sequenced"
## [5] "brca_tcga_cna" "brca_tcga_methylation_hm27"
## [7] "brca_tcga_methylation_hm450" "brca_tcga_mrna"
## [9] "brca_tcga_rna_seq_v2_mrna" "brca_tcga_rppa"
## [11] "brca_tcga_cnaseq"getGeneticProfiles(mycgds,mycancerstudy)[,1] ## [1] "brca_tcga_rppa" ## [2] "brca_tcga_rppa_Zscores" ## [3] "brca_tcga_protein_quantification" ## [4] "brca_tcga_protein_quantification_zscores" ## [5] "brca_tcga_gistic" ## [6] "brca_tcga_mrna" ## [7] "brca_tcga_mrna_median_Zscores" ## [8] "brca_tcga_rna_seq_v2_mrna" ## [9] "brca_tcga_rna_seq_v2_mrna_median_Zscores" ## [10] "brca_tcga_linear_CNA" ## [11] "brca_tcga_methylation_hm450" ## [12] "brca_tcga_mutations"
mycaselist ='brca_tcga_rna_seq_v2_mrna'
mygeneticprofile = 'brca_tcga_rna_seq_v2_mrna'
# Get data slices for a specified list of genes, genetic profile and case list
expr=getProfileData(mycgds,c('BRCA1','BRCA2'),mygeneticprofile,mycaselist)
DT::datatable(expr)很簡單就得到了指定基因在指定癌癥的表達量
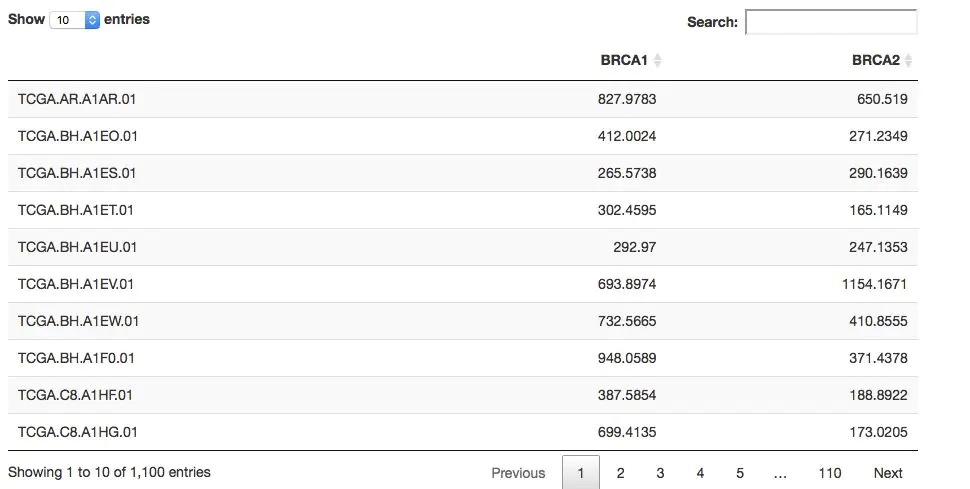
myclinicaldata = getClinicalData(mycgds,mycaselist) DT::datatable(myclinicaldata, extensions = 'FixedColumns', options = list( #dom = 't', scrollX = TRUE, fixedColumns = TRUE )) ## Warning in instance$preRenderHook(instance): It seems your data is too ## big for client-side DataTables. You may consider server-side processing: ## http://rstudio.github.io/DT/server.html
#突變基因名稱集合
mutGene=c("EGFR", "PTEN", "TP53", "ATRX")
#檢索基因和遺傳圖譜的基因組圖譜數據
mut_df <- getProfileData(mycgds,
caseList ="gbm_tcga_sequenced",
geneticProfile = "gbm_tcga_mutations",
genes = mutGene
)
mut_df <- apply(mut_df,2,as.factor)
mut_df[mut_df == "NaN"] = ""
mut_df[is.na(mut_df)] = ""
mut_df[mut_df != ''] = "MUT"
DT::datatable(mut_df)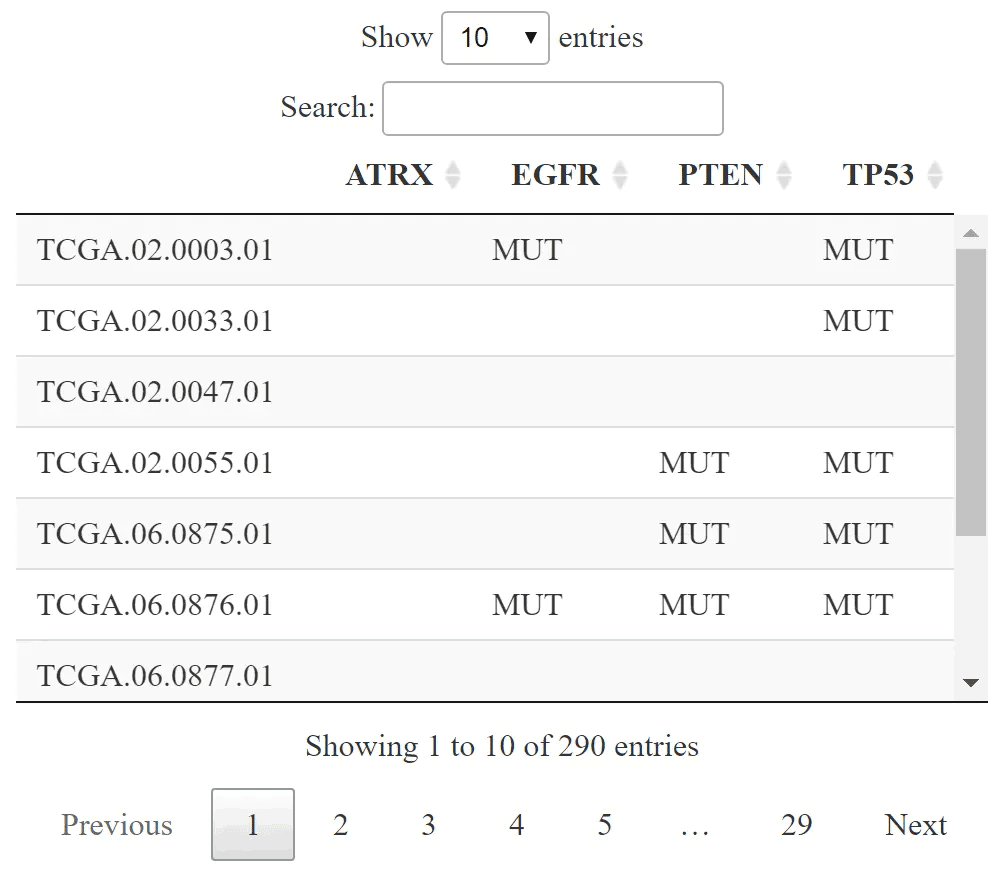
mutGene=c("TP53","UGT2B7","CYP3A4")
cna<-getProfileData(mycgds,mutGene,"gbm_tcga_gistic","gbm_tcga_sequenced")
cna<-apply(cna,2,function(x) as.character(factor(x,levels = c(-2:2),labels = c("HOMDEL","HETLOSS","DIPLOID","GAIN","AMP"))))
cna[is.na(cna)]=""
cna[cna=="DIPLOID"]=""
DT::datatable(cna)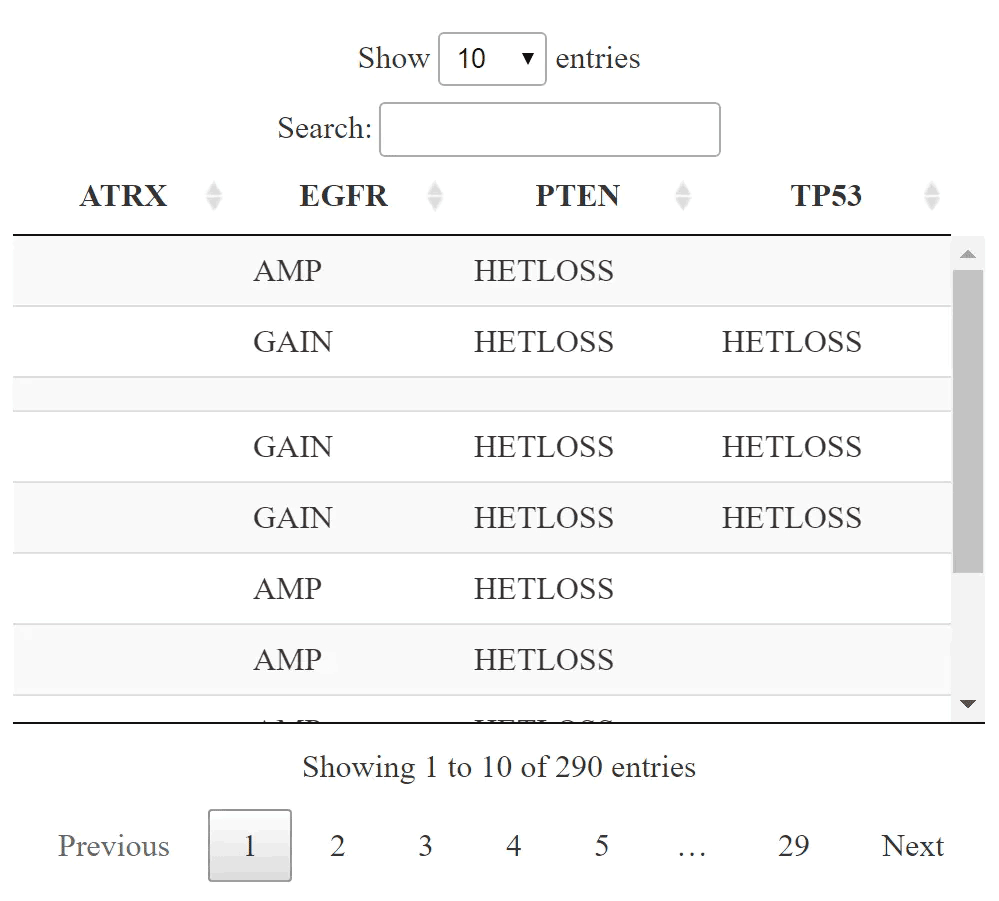
下面的函數,主要是配色比較復雜,其實原理很簡單,就是一個熱圖。
library(ComplexHeatmap)
library(grid)
conb <- data.frame(matrix(paste(as.matrix(cna),as.matrix(mut_df),sep = ";"), nrow=nrow(cna),ncol=ncol(cna), dimnames=list(row.names(mut_df),colnames(cna))))
mat <- as.matrix(t(conb))
DT::datatable((mat))
alt <- apply(mat,1,function(x)strsplit(x,";"))
alt <- unique(unlist(alt))
alt <- alt[which(alt !="")]
alt <-c("background",alt)
alter_fun = list( background = function(x,y,w,h){ grid.rect(x,y,w-unit(0.5,"mm"),h-unit(0.5,"mm"), gp=gpar(fill="#CCCCCC",col=NA)) }, HOMDEL = function(x,y,w,h){ grid.rect(x,y,w-unit(0.5,"mm"),h-unit(0.5,"mm"), gp=gpar(fill="blue3",col=NA)) }, HETLOSS = function(x,y,w,h){ grid.rect(x,y,w-unit(0.5,"mm"),h-unit(0.5,"mm"), gp=gpar(fill="cadetblue1",col=NA)) }, GAIN = function(x,y,w,h){ grid.rect(x,y,w-unit(0.5,"mm"),h-unit(0.5,"mm"), gp=gpar(fill="pink",col=NA)) }, AMP = function(x,y,w,h){ grid.rect(x,y,w-unit(0.5,"mm"),h-unit(0.5,"mm"), gp=gpar(fill="red",col=NA)) }, MUT = function(x,y,w,h){ grid.rect(x,y,w-unit(0.5,"mm"),h-unit(0.5,"mm"), gp=gpar(fill="#008000",col=NA)) })
col <- c("MUT"="#008000","AMP"="red","HOMDEL"="blue3", "HETLOSS"="cadetblue1","GAIN"="pink")
alt = intersect(names(alter_fun),alt)
alt_fun_list <- alter_fun[alt]
col <- col[alt]
oncoPrint(mat=mat,alter_fun = alt_fun_list, get_type = function(x) strsplit(x,";")[[1]], col = col)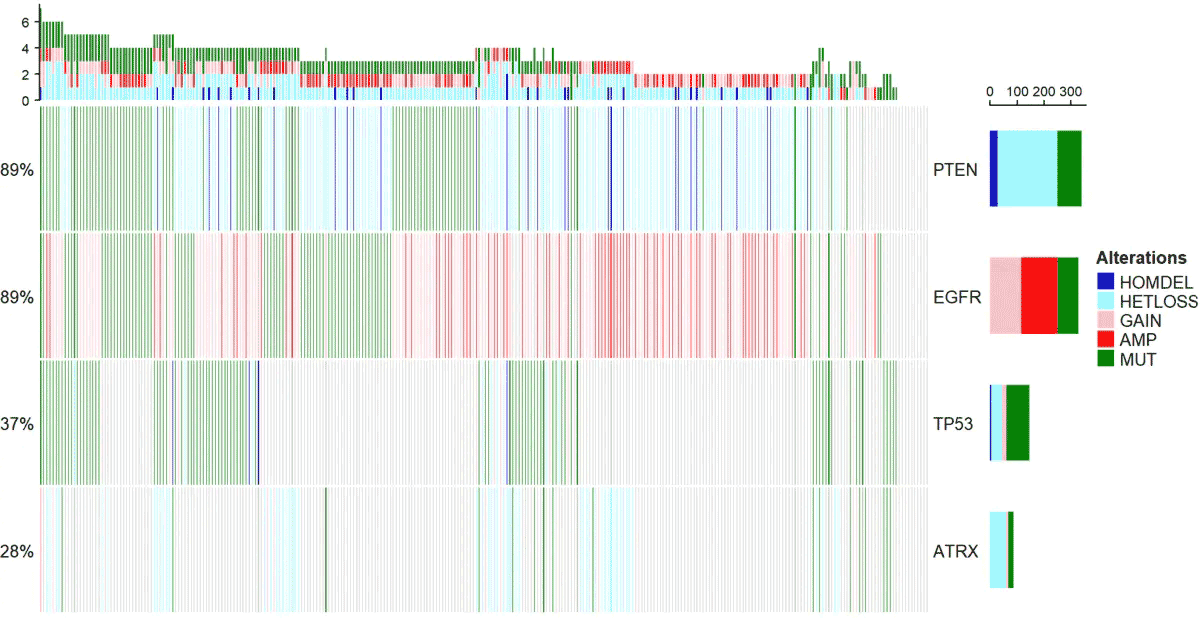
到此,相信大家對“R語言怎么使用cgdsr包獲取TCGA數據”有了更深的了解,不妨來實際操作一番吧!這里是億速云網站,更多相關內容可以進入相關頻道進行查詢,關注我們,繼續學習!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。