溫馨提示×
您好,登錄后才能下訂單哦!
點擊 登錄注冊 即表示同意《億速云用戶服務條款》
您好,登錄后才能下訂單哦!
如何使用R語言包circlize可視化展示blast雙序列比對結果,針對這個問題,這篇文章詳細介紹了相對應的分析和解答,希望可以幫助更多想解決這個問題的小伙伴找到更簡單易行的方法。
circlize這個包挺強大的,R語言里用來畫圈圖非常方便。
今天這篇文章記錄用circlize這個包畫圈圖展示blast雙序列比對結果的代碼
植物線粒體基因組類的文章通常會分析細胞器基因組間基因轉移情況,基本的分析方法就是blast比對。可視化展示可以選擇用這個圈圖來做
首先是使用blast建庫比對
makeblastdb -in mt.fasta -dbtype nucl -out mt
blastn -query cp.fasta -db mt -outfmt 6 > output.txt
然后是準備數據用來畫最外圈用來展示兩條序列的部分
df<-data.frame(chr=c(rep("chloroplast",2),rep("mitochondrial",2)),
x=c(1,131478,1,444567),
y=c(0,1,0,1))
> df
chr x y
1 chloroplast 1 0
2 chloroplast 131478 1
3 mitochondrial 1 0
4 mitochondrial 444567 1
然后是讀入blast的輸出結果
df1<-read.csv("output6.txt",stringsAsFactors = F,header=F,sep="\t")
作圖用到的代碼
library(circlize)
library(RColorBrewer)
library(ComplexHeatmap)
col<-RColorBrewer::brewer.pal(6,"Paired")
circos.par("start.degree" = 130)
circos.initialize(factors = df$chr,x=df$x)
circos.trackPlotRegion(factors = df$chr,y=df$y,
panel.fun = function(x,y){
circos.axis()
},track.height = 0.1)
highlight.sector(sector.index = "chloroplast",col=col[1])
highlight.sector(sector.index = "mitochondrial",col=col[2])
circos.text(x=70000,y=0.5,
labels = "chloroplast",
sector.index = "chloroplast")
circos.text(x=220000,y=0.5,
labels = "mitochondrial",
sector.index = "mitochondrial",
facing = "outside")
col_fun = colorRamp2(c(70,90,100),
c("green", "yellow", "red"))
for (i in 1:13){
x<-sort(c(df1[i,8],df1[i,7]))
y<-sort(c(df1[i,10],df1[i,9]))
z<-df1[i,3]
circos.link("chloroplast",x,"mitochondrial",y,
col=add_transparency(col_fun(z)))
}
circos.clear()
lgd_links = Legend(at = c(70, 80, 90, 100),
col_fun = col_fun,
title_position = "topleft",
title = "identity(%)")
lgd_list_vertical = packLegend(lgd_links)
draw(lgd_list_vertical, x = unit(10, "mm"),
y = unit(10, "mm"), just = c("left", "bottom"))
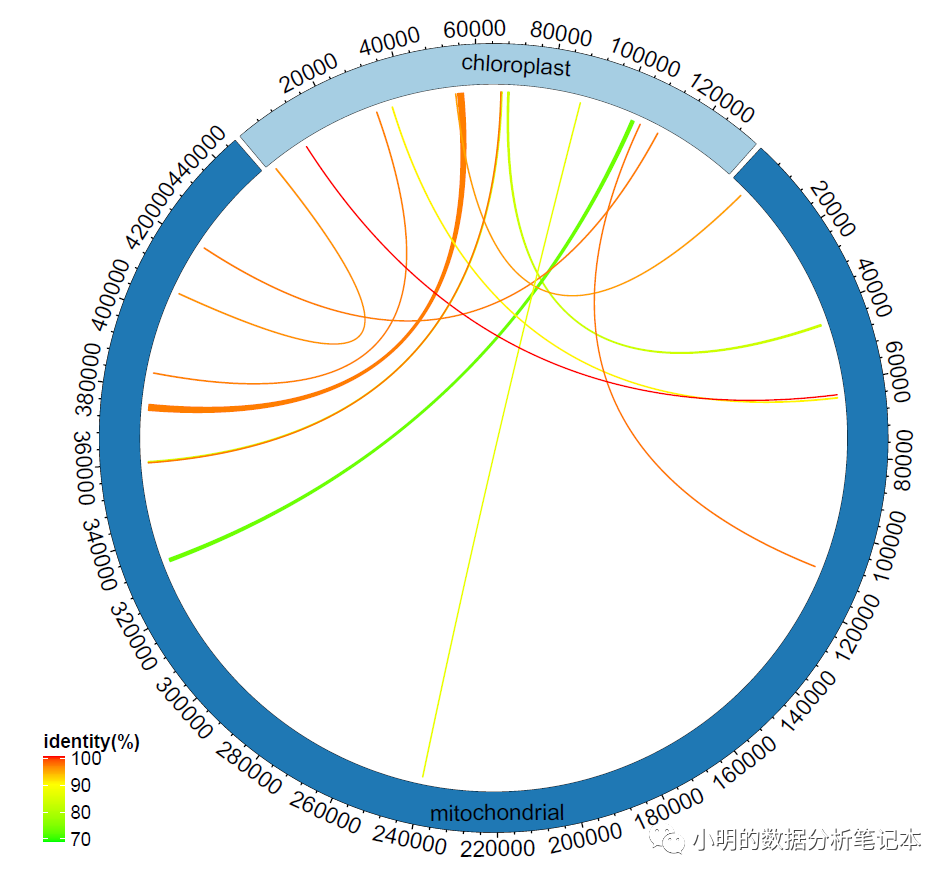
調整整體的角度
circos.par("start.degree" = 130)
調整用來表示染色體的外圈粗細
circos.trackPlotRegion(factors = df$chr,y=df$y,
panel.fun = function(x,y){
circos.axis()
},track.height = 0.1)
畫圖的時候可以加一個track.height參數
調整外圈的刻度,現在展示的有點多,我想增大間隔,減少展示的數字,暫時不知道如何實現。
關于如何使用R語言包circlize可視化展示blast雙序列比對結果問題的解答就分享到這里了,希望以上內容可以對大家有一定的幫助,如果你還有很多疑惑沒有解開,可以關注億速云行業資訊頻道了解更多相關知識。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。