您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
小編給大家分享一下R語言中如何進行多元邏輯回歸,希望大家閱讀完這篇文章之后都有所收獲,下面讓我們一起去探討吧!
如何進行多元邏輯回歸
可以使用階梯函數通過逐步過程確定多元邏輯回歸。此函數選擇模型以最小化AIC。
通常建議不要盲目地遵循逐步程序,而是要使用擬合統計(AIC,AICc,BIC)比較模型,或者根據生物學或科學上合理的可用變量建立模型。
多元相關是研究潛在自變量之間關系的一種工具。例如,如果兩個獨立變量彼此相關,可能在最終模型中都不需要這兩個變量,但可能有理由選擇一個變量而不是另一個變量。
創建數值變量的數據框
Data.num $ Status = as.numeric(Data.num $ Status) Data.num $ Length = as.numeric(Data.num $ Length) Data.num $ Migr = as.numeric(Data.num $ Migr) Data.num $ Insect = as.numeric(Data.num $ Insect) Data.num $ Diet = as.numeric(Data.num $ Diet) Data.num $ Broods = as.numeric(Data.num $ Broods) Data。 num $ Wood = as.numeric(Data.num $ Wood) Data.num $ Upland = as.numeric(Data.num $ Upland) Data.num $ Water = as.numeric(Data.num $ Water) Data.num $ Release = as.numeric(Data.num $ Release) Data.num $ Indiv = as.numeric(Data.num $ Indiv) ###檢查新數據框 headtail(Data.num) 1 1 1520 9600.0 1.21 1 12 2 6.0 1 0 0 1 6 29 2 1 1250 5000.0 0.56 1 0 1 6.0 1 0 0 1 10 85 3 1 870 3360.0 0.07 1 0 1 4.0 1 0 0 1 3 8 77 0 170 31.0 0.55 3 12 2 4.0 NA 1 0 0 1 2 78 0 210 36.9 2.00 2 8 2 3.7 1 0 0 1 1 2 79 0 225 106.5 1.20 2 12 2 4.8 2 0 0 0 1 2 ###檢查變量之間的相關性 ###這里使用了Spearman相關性

在此示例中,數據包含缺失值。在R中缺失值用NA表示。SAS通常會無縫地處理缺失值。雖然這使用戶更容易,但可能無法確保用戶了解這些缺失值的作用。在某些情況下,R要求用戶明確如何處理缺失值。處理多元回歸中的缺失值的一種方法是從數據集中刪除具有任何缺失值的所有觀察值。這是我們在逐步過程之前要做的事情,創建一個名為Data.omit的數據框。但是,當我們創建最終模型時,我們只想排除那些在最終模型中實際包含的變量中具有缺失值的觀察。為了測試最終模型的整體p值,繪制最終模型,或使用glm.compare函數,我們將創建一個名為Data.final的數據框,只排除那些觀察結果。
盡管二項式和poission系列中的模型應該沒問題,但是對于使用某些glm擬合的步驟過程存在一些注意事項。
最終模型
summary(model.final) Coefficients: Estimate Std. Error z value Pr(>|z|) (Intercept) -3.5496482 2.0827400 -1.704 0.088322 . Upland -4.5484289 2.0712502 -2.196 0.028093 * Migr -1.8184049 0.8325702 -2.184 0.028956 * Mass 0.0019029 0.0007048 2.700 0.006940 ** Indiv 0.0137061 0.0038703 3.541 0.000398 *** Insect 0.2394720 0.1373456 1.744 0.081234 . Wood 1.8134445 1.3105911 1.384 0.166455
偽R方
$Pseudo.R.squared.for.model.vs.null Pseudo.R.squared McFadden 0.700475 Cox and Snell (ML) 0.637732 Nagelkerke (Cragg and Uhler) 0.833284
在最終模型中創建包含變量的數據框,并省略NA。
Analysis of Deviance Table Model 1: Status ~ Upland + Migr + Mass + Indiv + Insect + Wood Model 2: Status ~ 1 Resid. Df Resid. Dev Df Deviance Pr(>Chi) 1 63 30.392 2 69 93.351 -6 -62.959 1.125e-11 ***
Likelihood ratio test #Df LogLik Df Chisq Pr(>Chisq) 1 7 -15.196 2 1 -46.675 -6 62.959 1.125e-11 ***
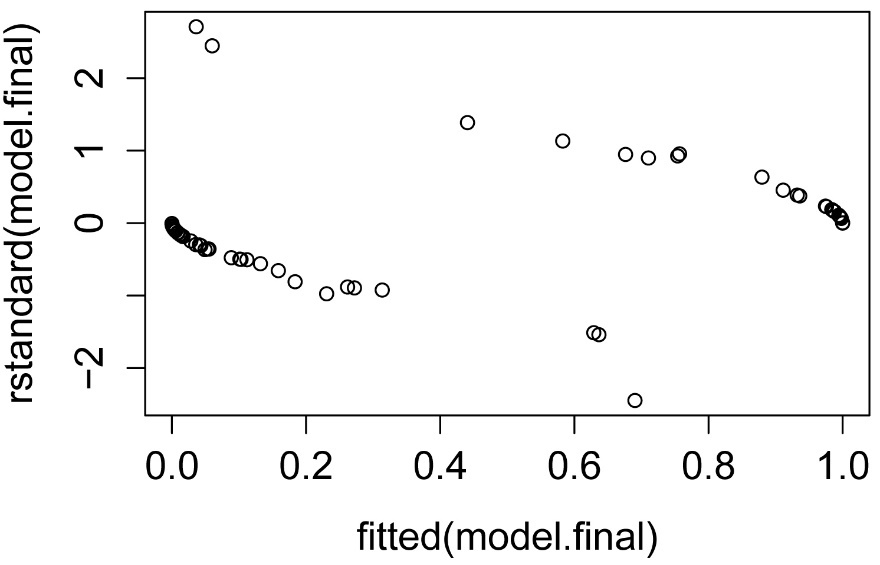
在最終模型中創建包含變量的數據框,并在NA中省略
過度離散是glm的deviance殘差相對于自由度較大的情況。這些值顯示在模型的摘要中。一個指導原則是,如果deviance殘差與剩余自由度的比率超過1.5,則模型過度離散。過度離散表明模型不能很好地擬合數據:解釋變量可能無法很好地描述因變量,或者可能無法為這些數據正確指定模型。如果存在過度離散,一種可能的解決方案是 在glm中使用quasibinomial family選項。
Null deviance: 93.351 on 69 degrees of freedom Residual deviance: 30.392 on 63 degrees of freedom deviance / df.residual [1] 0.482417
使用逐步程序的替代或補充是將模型與擬合統計進行比較。我的compare.glm 函數將為glm模型顯示AIC,AICc,BIC和偽R平方。使用的模型應該都擬合相同的數據。也就是說,如果數據集中的不同變量包含缺失值,則應該謹慎使用。如果您對使用哪種擬合統計數據沒有任何偏好,您希望在最終模型中使用較少的術語,我可能會推薦AICc或BIC。
一系列模型可以與標準的anova 功能進行比較。模型應嵌套在先前模型中或anova函數列表中的下一個模型中; 和模型應該擬合相同的數據。在比較多個回歸模型時,通常放寬p值為0.10或0.15。
在以下示例中,使用通過逐步過程選擇的模型。請注意,雖然模型9最小化了AIC和AICc,但模型8最小化了BIC。anova結果表明模型8不是對模型7的顯著改進。這些結果支持選擇模型7,8或9中的任何一個。
compareGLM(model.1, model.2, model.3, model.4, model.5, model.6, model.7, model.8, model.9) $Models Formula 1 "Status ~ 1" 2 "Status ~ Release" 3 "Status ~ Release + Upland" 4 "Status ~ Release + Upland + Migr" 5 "Status ~ Release + Upland + Migr + Mass" 6 "Status ~ Release + Upland + Migr + Mass + Indiv" 7 "Status ~ Release + Upland + Migr + Mass + Indiv + Insect" 8 "Status ~ Upland + Migr + Mass + Indiv + Insect" 9 "Status ~ Upland + Migr + Mass + Indiv + Insect + Wood" $Fit.criteria Rank Df.res AIC AICc BIC McFadden Cox.and.Snell Nagelkerke p.value 1 1 66 94.34 94.53 98.75 0.0000 0.0000 0.0000 Inf 2 2 65 62.13 62.51 68.74 0.3787 0.3999 0.5401 2.538e-09 3 3 64 56.02 56.67 64.84 0.4684 0.4683 0.6325 3.232e-10 4 4 63 51.63 52.61 62.65 0.5392 0.5167 0.6979 7.363e-11 5 5 62 50.64 52.04 63.87 0.5723 0.5377 0.7263 7.672e-11 6 6 61 49.07 50.97 64.50 0.6118 0.5618 0.7588 5.434e-11 7 7 60 46.42 48.90 64.05 0.6633 0.5912 0.7985 2.177e-11 8 6 61 44.71 46.61 60.14 0.6601 0.5894 0.7961 6.885e-12 9 7 60 44.03 46.51 61.67 0.6897 0.6055 0.8178 7.148e-12 Analysis of Deviance Table Model 1: Status ~ 1 Model 2: Status ~ Release Model 3: Status ~ Release + Upland Model 4: Status ~ Release + Upland + Migr Model 5: Status ~ Release + Upland + Migr + Mass Model 6: Status ~ Release + Upland + Migr + Mass + Indiv Model 7: Status ~ Release + Upland + Migr + Mass + Indiv + Insect Model 8: Status ~ Upland + Migr + Mass + Indiv + Insect Model 9: Status ~ Upland + Migr + Mass + Indiv + Insect + Wood Resid. Df Resid. Dev Df Deviance Pr(>Chi) 1 66 90.343 2 65 56.130 1 34.213 4.94e-09 *** 3 64 48.024 1 8.106 0.004412 ** 4 63 41.631 1 6.393 0.011458 * 5 62 38.643 1 2.988 0.083872 . 6 61 35.070 1 3.573 0.058721 . 7 60 30.415 1 4.655 0.030970 * 8 61 30.710 -1 -0.295 0.587066 9 60 28.031 1 2.679 0.101686
看完了這篇文章,相信你對“R語言中如何進行多元邏輯回歸”有了一定的了解,如果想了解更多相關知識,歡迎關注億速云行業資訊頻道,感謝各位的閱讀!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。