溫馨提示×
您好,登錄后才能下訂單哦!
點擊 登錄注冊 即表示同意《億速云用戶服務條款》
您好,登錄后才能下訂單哦!
今天小編給大家分享一下GWAS數字協變量怎么用的相關知識點,內容詳細,邏輯清晰,相信大部分人都還太了解這方面的知識,所以分享這篇文章給大家參考一下,希望大家閱讀完這篇文章后有所收獲,下面我們一起來了解一下吧。
第一列為FID 第二列為ID 第三列以后為協變量(注意,只能是數字,不能是字符!)
這里協變量文件為:
[dengfei@ny 03_linear_cov]$ head cov.txt
1061 1061 F 3
1062 1062 M 3
1063 1063 F 3
1064 1064 F 3
1065 1065 F 3
1066 1066 F 3
1067 1067 F 3
1068 1068 M 3
1069 1069 M 3
1070 1070 M 3
這里第三列為性別,第四列為世代,為了方便操作,我們將世代作為數值,直接進行協變量分析
awk '{print $1,$2,$4}' cov.txt >cov1.txt
數據如下:
1061 1061 3
1062 1062 3
1063 1063 3
1064 1064 3
1065 1065 3
1066 1066 3
1067 1067 3
1068 1068 3
1069 1069 3
1070 1070 3
「代碼:」
plink --file b --pheno phe.txt --allow-no-sex --linear --covar cov1.txt --out re
「日志:」
PLINK v1.90b5.3 64-bit (21 Feb 2018) www.cog-genomics.org/plink/1.9/
(C) 2005-2018 Shaun Purcell, Christopher Chang GNU General Public License v3
Logging to re.log.
Options in effect:
--allow-no-sex
--covar cov1.txt
--file b
--linear
--out re
--pheno phe.txt
515199 MB RAM detected; reserving 257599 MB for main workspace.
.ped scan complete (for binary autoconversion).
Performing single-pass .bed write (10000 variants, 1500 people).
--file: re-temporary.bed + re-temporary.bim + re-temporary.fam written.
10000 variants loaded from .bim file.
1500 people (0 males, 0 females, 1500 ambiguous) loaded from .fam.
Ambiguous sex IDs written to re.nosex .
1500 phenotype values present after --pheno.
Using 1 thread (no multithreaded calculations invoked).
--covar: 1 covariate loaded.
Before main variant filters, 1500 founders and 0 nonfounders present.
Calculating allele frequencies... done.
10000 variants and 1500 people pass filters and QC.
Phenotype data is quantitative.
Writing linear model association results to re.assoc.linear ... done.
「結果文件:」re.assoc.linear
「結果預覽:」
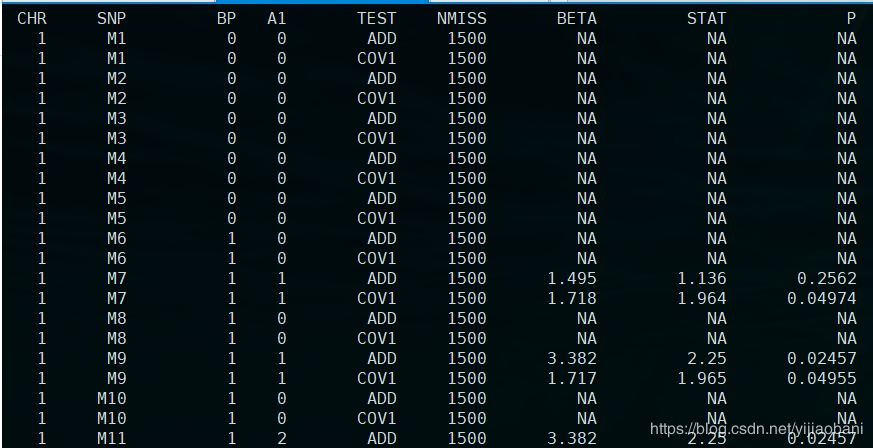
這里的結果包括協變量的檢驗,我們不需要輸出協變量結果,可以加上參數:--hide-covar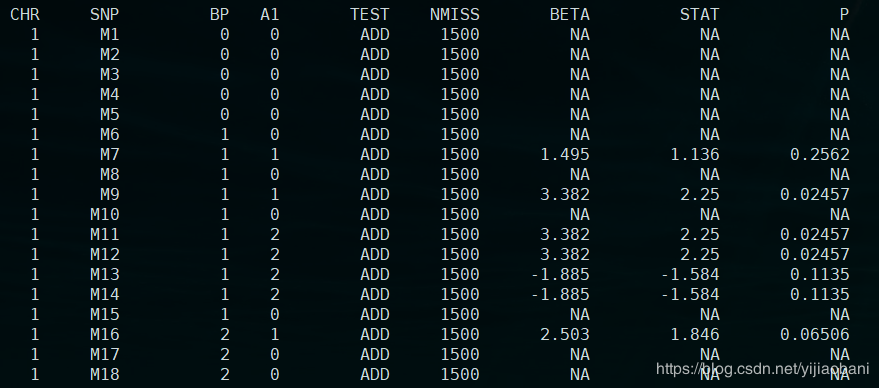
library(data.table)
geno = fread("c.raw")
geno[1:10,1:10]
phe = fread("phe.txt")
cov = fread("cov.txt")
dd = data.frame(phe$V3,cov$V4,geno[,7:20])
head(dd)
str(dd)
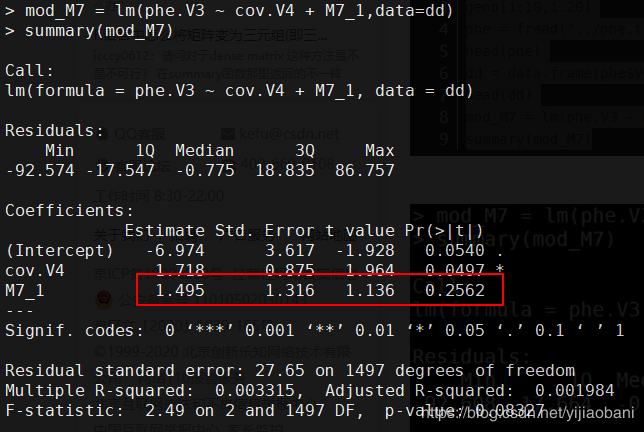
mod_M7 = lm(phe.V3 ~ cov.V4 + M7_1,data=dd)
summary(mod_M7)
mod_M9 = lm(phe.V3 ~ cov.V4 + M9_1,data=dd);summary(mod_M9)
M7加上數值協變量結果:
M9加上數值協變量結果:
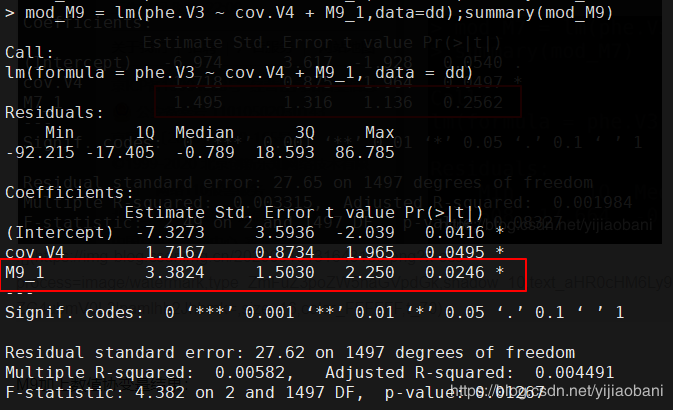
結果完全一致。
以上就是“GWAS數字協變量怎么用”這篇文章的所有內容,感謝各位的閱讀!相信大家閱讀完這篇文章都有很大的收獲,小編每天都會為大家更新不同的知識,如果還想學習更多的知識,請關注億速云行業資訊頻道。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。