您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章主要講解了“Trimmomatic怎么使用”,文中的講解內容簡單清晰,易于學習與理解,下面請大家跟著小編的思路慢慢深入,一起來研究和學習“Trimmomatic怎么使用”吧!
二代測序數據的指控一般包含以下步驟:
切除尾端堿基質量小于指定值(一般為20)的堿基。可以簡單的單堿基修剪,也即從末端開始進行刪除,直到讀取堿基質量高于20;也可以進行滑窗修剪,也即從末端開始以指定堿基數目的滑窗開始修剪,直到滑窗內堿基平均質量高于20。
去除末端修剪后長度小于指定值的reads。不同項目指定值不同,一般宏基因組去掉小于50bp的reads(50bp已不夠產生k-mer),而擴增子測序則根據raw reads長度和PCR插入片段的長度來確定,例如V4區大概260bp,那么可以去掉雙末端reads之和小于280bp的(否則不足以拼接)。
其他一些要求,例如去除含有N(也即無法讀取位點)過多的reads、去除完全重復的reads等。
conda install -c trimmomatic
java -jar trimmomatic-0.30.jar PE -threads 20 -phred33 R1.fq R2.fq clean.R1.fq unpaired.R1.fq clean.R2.fq unpaired.R2.fq ILLUMINACLIP:TruSeq3-PE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:36參數解釋如下:PE/SE 設定對Paired-End或Single-End的reads進行處理,其輸入和輸出參數稍有不一樣。-threads 設置多線程運行數,也即核數-phred33 設置堿基的質量格式,可選pred64ILLUMINACLIP:TruSeq3-PE.fa:2:30:10 切除adapter序列。參數后面分別接adapter序列的fasta文件:允許的最大mismatch數:palindrome模式下匹配堿基數閾值:simple模式下的匹配堿基數閾值。LEADING:3 切除首端堿基質量小于3的堿基TRAILING:3 切除尾端堿基質量小于3的堿基SLIDINGWINDOW:4:15 滑窗修剪,一個Windows的size是4個堿基,其平均堿基質量小于15,則切除。MINLEN:50 最小的reads長度CROP:<length> 保留reads到指定的長度HEADCROP:<length> 在reads的首端切除指定的長度TOPHRED33 將堿基質量轉換為pred33格式TOPHRED64 將堿基質量轉換為pred64格式
切除尾端堿基質量小于20的堿基(也即從末端開始進行刪除,直到讀取堿基質量高于20),并去掉剪切后長度小于150的小序列片段:java -jar trimmomatic-0.30.jar PE -threads 20 -phred33 R1.fq R2.fq clean.R1.fq unpaired.R1.fq clean.R2.fq unpaired.R2.fq TRAILING:20 MINLEN:150使用末端滑窗修剪,同時去掉質控后長度過短(小于50bp)的小片段,如下所示:java -jar trimmomatic-0.33.jar PE -threads 20 -phred33 rm_dup_N_trim_1.fq rm_dup_N_trim_2.fq clean_1.fq unp_clean_1.fq clean_2.fq unp_clean_2.fq SLIDINGWINDOW:4:20 MINLEN:50
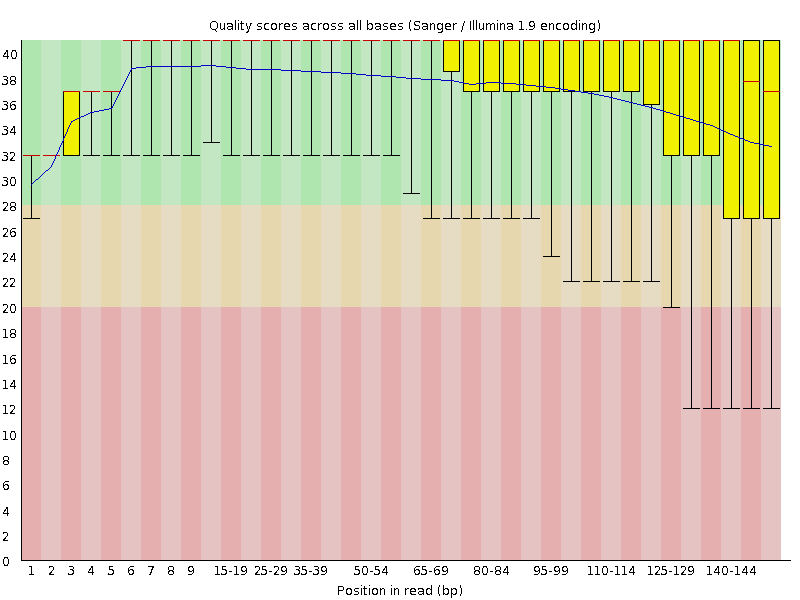
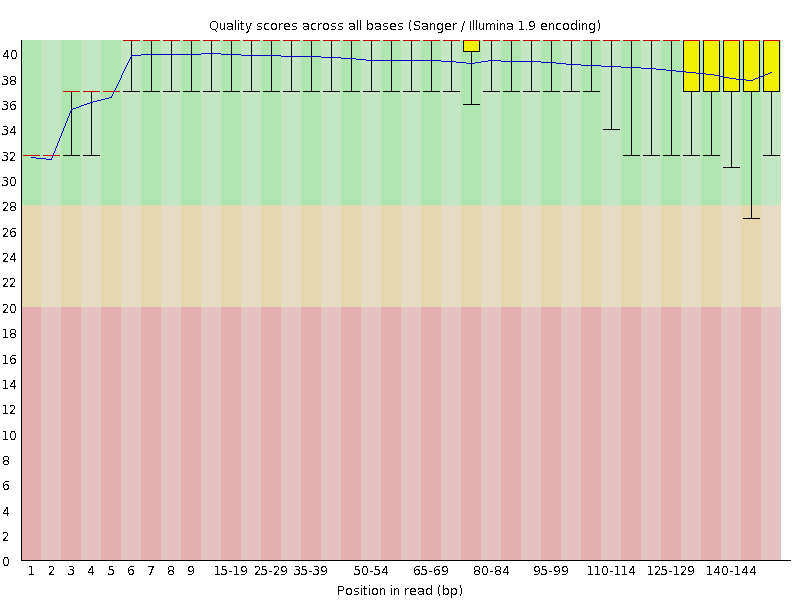
感謝各位的閱讀,以上就是“Trimmomatic怎么使用”的內容了,經過本文的學習后,相信大家對Trimmomatic怎么使用這一問題有了更深刻的體會,具體使用情況還需要大家實踐驗證。這里是億速云,小編將為大家推送更多相關知識點的文章,歡迎關注!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。