您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
怎么使用FitHiC評估染色質交互作用的顯著性,相信很多沒有經驗的人對此束手無策,為此本文總結了問題出現的原因和解決方法,通過這篇文章希望你能解決這個問題。
通過Hi-C技術可以得到全基因組范圍內的染色質交互信息, 在不同的分辨率下,首先得到bin之間的交互矩陣contact matrix, 通過熱圖的形式來展示該交互矩陣,即得到了contact map。在完整的contac matrix的基礎上,可以進行A/B隔室,拓撲結構域,染色質環等不同層級空間結構單元的分析。
正是由于覆蓋了全基因組范圍內的染色質交互信息,才使得hi-C技術可以站在全基因組的高度對不同層級的空間結構進行挖掘,這個是hi-c技術獨有的優勢。作為3C技術的升級版,hi-c也是可以直接研究某些染色質之間的交互作用的,只不過由于測序和序列比對等系統誤差的存在,在交互矩陣中還是有部分信息是不可靠的,為了通過hi-c技術來直接分析某些染色質之間的互作,科學家發明了很多的算法,對交互矩陣中的信息進行評估,通過打分等形式來提取限制性的交互信息,而FitHic就是其中最常用的一款軟件。
該軟件最初采用python進行開發,后來為了使用方便,將相關功能重寫并封裝成了一個R包。
該軟件的原理示意如下
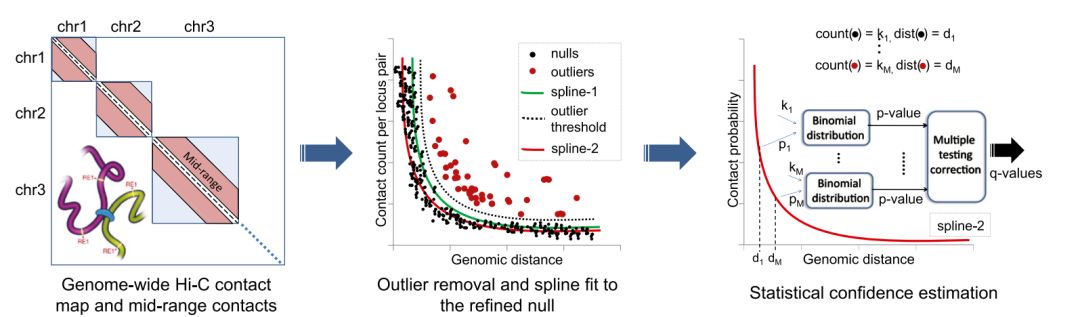
從原始的交互矩陣中,根據事先定義的距離閾值提取出mid-range,即中等距離的同一個染色質bin之間的交互作用。在文章中指出,對于酵母,中等距離的范圍為10kb到25kb, 對于人和小鼠,中等距離的范圍為50kb到10Mb, 這里的距離為兩個bin之間的線性距離。
根據提取出的mid-range交互信息,首先構建基因組線性距離與交互頻率的模型,即圖中的spline-1, 在該模型的基礎上制定過濾的閾值,即虛線代表的outlier-threshold, 然后提出離群值數據,對應圖中的紅色原點。對于剩下的數據再次進行擬合,得到spline2。 然后在二項分布的基礎上計算每個交互作用的pvalue,再進行多種假設檢驗的校正,得到qvalue。
該軟件的用法簡單,只不過需要對原始的交互矩陣進行格式化。一個經典的交互矩陣如下所示
Bin1 Bin2 Bin3 Bin4 Bin5 Bin6
7.85957 4.80329 11.4766 9.57416 4.5288 8.55022
8.61621 4.98956 2.35654 5.69483 11.1187 10.1322
4.06803 4.07801 7.98047 2.59144 6.3851 7.74306
4.52869 2.70624 8.94544 4.29185 8.29491 8.38257每一行和每一列都代表一個bin, 數字代表兩個bin之間的交互頻率。在此文件的基礎上,經過如下兩步即可得到顯著性評估的結果。
該軟件至少需要準備兩個輸入文件,第一個文件為bin對應的染色質區域,稱之為fragsfile, 內容示意如下
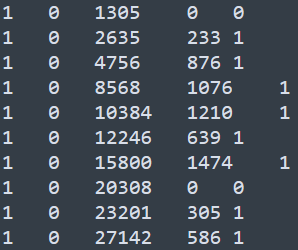
\t分隔的5列,其中第二列和第五列的信息沒有作用,用0或者1填充就可以了,第一列表示bin所在的染色體,第三列代表bin的中心位置, 第三列代表與該bin存在交互的頻率總和,即交互矩陣中對應列或者行的總和。
第二個文件為bin之間交互頻率的信息,稱之為intersfile, 內容示意如下
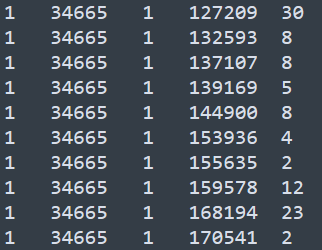
\t分隔的5列,前兩列代表第一個bin的染色質名稱和中心位置,第三列和第四列代表第二個bin的染色質名稱和中心位置,第五列代表兩個bin之間的交互頻率。
準備好輸入文件之后,就可以運行了,基本用法如下
FitHiC(
fragsfile,
intersfile,
outdir,
libname = "test_project",
distUpThres = 250000,
distLowThres = 10000,
visual = TRUE)指定兩個輸入文件和輸出結果的目錄,libname指定輸出文件的前綴,distUpThres和distLowThres指定距離的上下閾值,以此閾值來篩選得到mid-range。
在輸出結果中所有文件分成了pass1和pass2兩個部分,每個部分有對應的以下4張圖
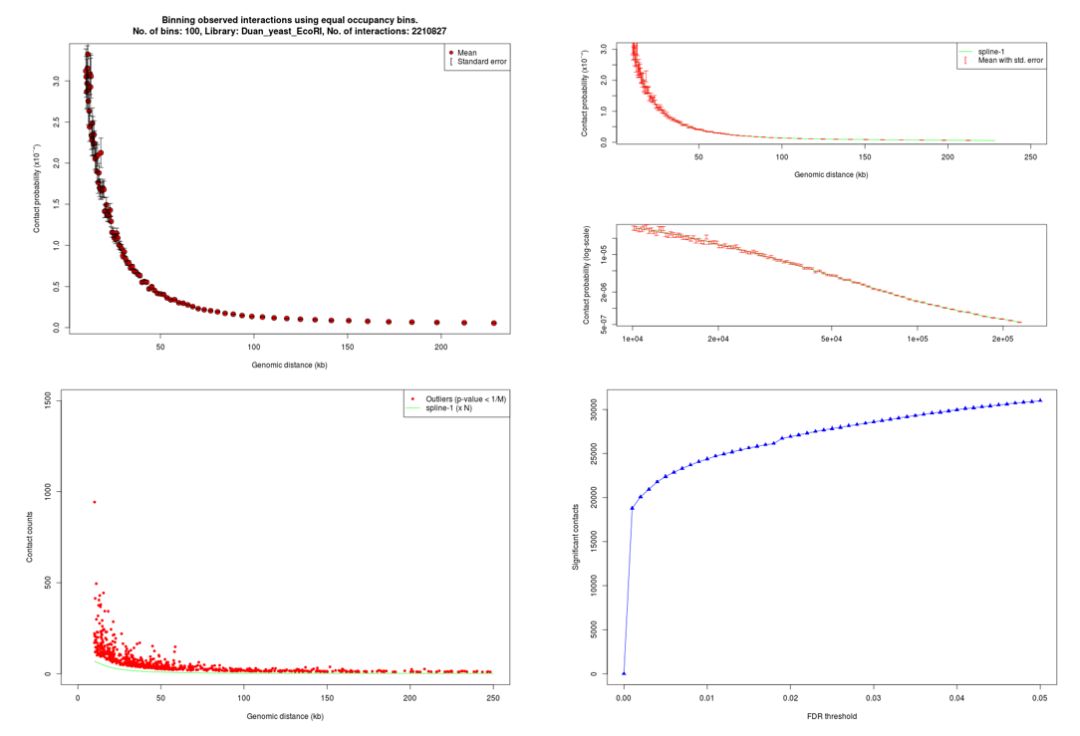
第一張圖表示基于mid-range的交互信息得到的基因組線性距離與交互概率的分布,第二張圖表示擬合得到的分布,第三張圖表示擬合模型篩選得到的離群值,第四張圖表示不同FDR閾值篩選的顯著交互作用的分布。
最終得到的顯著性評估結果可以從后綴為pass2.significances.txt.gz的文件中得到,該文件內容示意如下
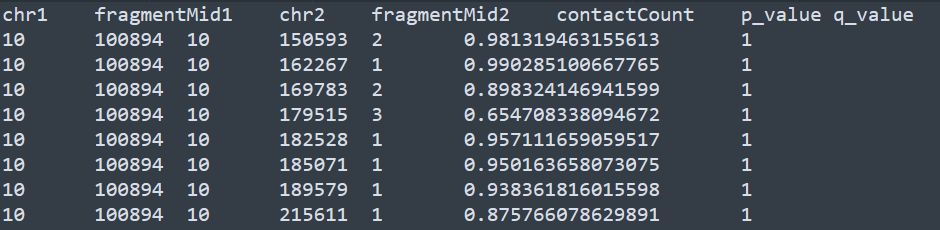
通過最后一列的qvaue作為閾值,去篩選得到顯著性的染色質互作。
看完上述內容,你們掌握怎么使用FitHiC評估染色質交互作用的顯著性的方法了嗎?如果還想學到更多技能或想了解更多相關內容,歡迎關注億速云行業資訊頻道,感謝各位的閱讀!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。