您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
小編給大家分享一下CPAT軟件有什么用,希望大家閱讀完這篇文章之后都有所收獲,下面讓我們一起去探討吧!
隨著高通量測序在lncRNA研究領域的應用, 越來越多的lncRNA被發現。對于轉錄組測序的數據而言,組裝得到轉錄本之后,首先要做的就是區分蛋白編碼和非蛋白編碼的RNA。
目前針對這一問題,有多種解決方案,基本可以分為以下兩類
alignment-based
alignment-free
第一種算法基于序列比對,可以較好的識別保守性較好的蛋白編碼基因, 包括CPC,PhyloCSF等軟件; 第二種算法不需要比對,而是通過coding和non-coding轉錄本的序列特征來進行區分,包括CNCI, CPAT, PLEK等。
lncRNA在物種間的保守性較差,另外部分lncRNA的染色體位置和蛋白編碼基因存在重疊,通過序列比對的方式來區分容易造成誤判。除此之外,基于序列比對的軟件,其運行速度相對較慢,所以采用第二種算法的軟件綜合效果更好。
本文主要介紹CPAT的使用,網址如下
http://lilab.research.bcm.edu/cpat/
對于一個轉錄本而言,它是coding還是noncoding, 本質上是一個二分類問題,所以CPAT的開發者想到了通過邏輯回歸來解決這個問題。該軟件基于以下四個特征構建了邏輯回歸模型來區分coding和noncoding
open reading frame size
open reading frame coverage
Fickett TESTCODE statistic
hexamer usage bias
前兩個因素都是針對開放閱讀框定義的,第一個因素是開放閱讀框的大小,第二個因素是開放閱讀框占轉錄本總長度的比例,第三個因素基于序列的堿基組成和密碼子分布進行定義,第四個因素基于序列中六聚體的頻率進行定義。
在論文中,針對以上4種特征,首先評估在coding和noncoding中的分布,圖示如下
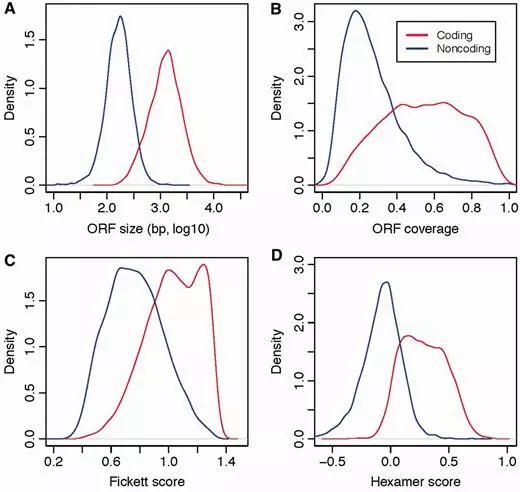
可以看到,coding和noncoding形成了兩個不同的峰,說明這4種特征在coding和noncoding之間確實存在差異。
在論文中還通過ROC曲線評估了不同軟件的性能,結果如下
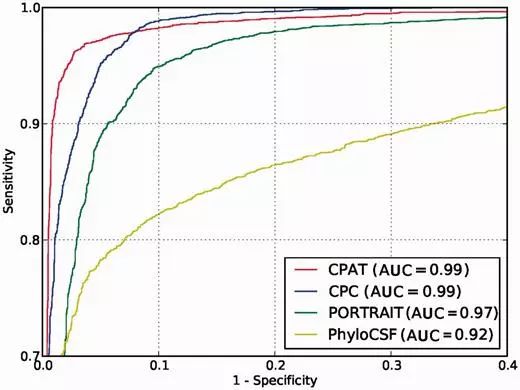
可以看到CPAT和CPC的效果是最好的。CPAT基于python編程語言開發,安裝非常的簡便,代碼如下
pip install CPAT
該軟件既可以在本地運行,也提供了在線版本。
在線版本的網址如下
http://lilab.research.bcm.edu/cpat/
可以直接輸入fasta格式的序列,也可以輸入bed格式的文件,此時需要指定對應的基因組版本,示意如下
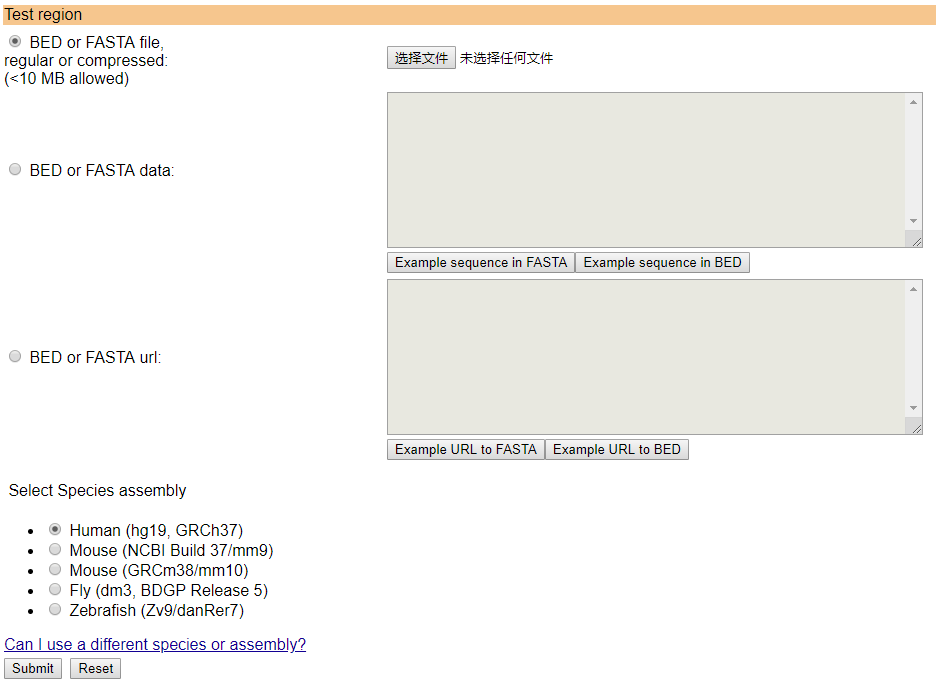
本地版本對應的也有兩種用法,輸入bed文件的用法如下
cpat.py -r /database/hg19.fa \ -g mRNA_hg19.bed \ -d dat/Human_logitModel.RData \ -x dat/Human_Hexamer.tsv \ -o output.txt
輸入fasta文件的用法如下
cpat.py -g transcript.fa \ -d dat/Human_logitModel.RData \ -x dat/Human_Hexamer.tsv \ -o output.txt
-d和-x參數對應的文件為軟件構建好的模型,位于軟件的安裝目錄下。軟件的輸出結果如下所示

最后一列給出了轉錄本的蛋白編碼信息,yes代表該轉錄本為protein-coding轉錄本,no代表該轉錄本為noncoding轉錄本。
看完了這篇文章,相信你對“CPAT軟件有什么用”有了一定的了解,如果想了解更多相關知識,歡迎關注億速云行業資訊頻道,感謝各位的閱讀!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。