您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
怎么使用trim_galore對NGS數據進行質量過濾,相信很多沒有經驗的人對此束手無策,為此本文總結了問題出現的原因和解決方法,通過這篇文章希望你能解決這個問題。
cutadapt軟件可以對NGS數據進行質量過濾,FastQC軟件可以查看NGS數據的質量分布,trim_galore將這兩個軟件封裝到一起,使用起來更加的方便。
該軟件會對數據進行以下4步處理
illumina平臺的測序數據,通常3’端質量較差。trim_galore首先會過濾掉3’端的低質量堿基,本質上是調用了cutadapt的質量過濾算法。下圖是過濾前后堿基質量的分布圖
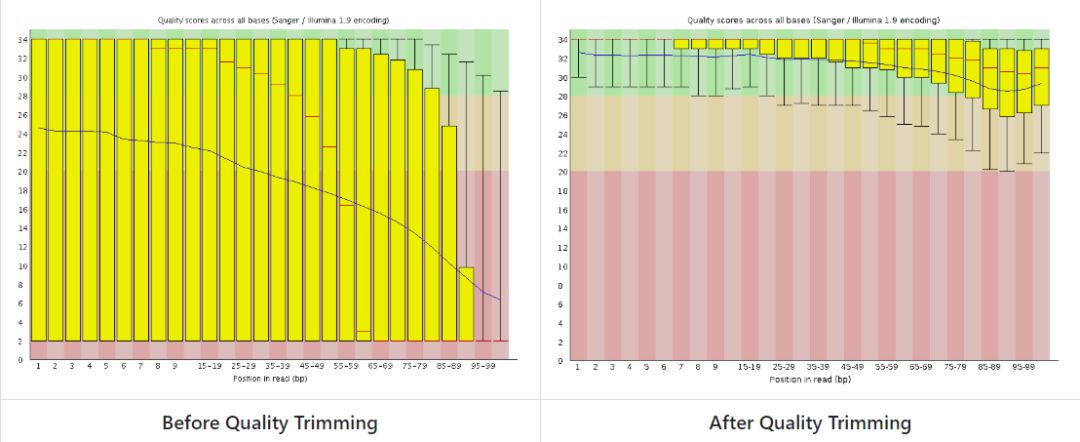
可以看到,過濾掉低質量堿基后,序列的整體質量顯著提高。
過濾掉低質量的堿基之后,trim_galore會調用cutadapt在reads的3’端查找adapter 序列并去除。通常情況下,我們需要指定對應的adapter序列,如果沒有指定的化,trim_galore會自動查找以下3種類型的adapter
Illumina: AGATCGGAAGAGC Small RNA: TGGAATTCTCGG Nextera: CTGTCTCTTATA
默認讀取前一百萬條序列,通過這一百萬條序列判斷adapter屬于上述三種的哪一種,然后進行去除。如果你不希望軟件自動判斷,也可以通過--illumina, --nextera, --small_rna參數指定對應的adapter類型。
經過上述兩步處理之后,有可能剩余的序列長度很短,這部分短序列也會被去除。默認情況下,如果序列長度少于20bp, 這條序列會被丟掉。
對于所有的輸入序列,以上3個步驟是肯定會執行的。除此之,trim_galore還支持一些其他的過濾措施,以滿足個性化的需求。
hardtrim5參數用于從序列的3’端切除堿基,示意如下
before: CCTAAGGAAACAAGTACACTCCACACATGCATA --hardtrim5 20: CCTAAGGAAACAAGTACACT
通過hardtrim5參數可以將序列截取成固定長度。與之對應的,還有一個hardtrim3參數,從序列的5’端切除堿基,示意如下
before: CAAATGTTATTTTTAAGAAAATGGAAAAT --hardtrim3 20: TTTTTAAGAAAATGGAAAAT
軟件的安裝也很方便,首先需要確保cutadapt和fastqc這兩個軟件已經安裝,并且可執行文件位于PAH環境變量定義的路徑種。然后下載trim_galore的源代碼包,解壓即可,代碼如下
wget https://github.com/FelixKrueger/TrimGalore/archive/0.5.0.tar.gz tar xzvf 0.5.0.tar.gz
在軟件的安裝目錄有一個名為trim_galore的可執行文件。
對于單端測序數據,基本用法如下
trim_galore --quality 20 -a AGATCGGAAGAGC --length 20 -o out_dir input.fq
對于雙端測序數據,基本用法如下
trim_galore --paired --quality 20 -a AGATCGGAAGAGC -a2 AGATCGGAAGAGC --length 20 -o out_dir R1.fq.gz R2.fq.gz
看完上述內容,你們掌握怎么使用trim_galore對NGS數據進行質量過濾的方法了嗎?如果還想學到更多技能或想了解更多相關內容,歡迎關注億速云行業資訊頻道,感謝各位的閱讀!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。